


随着基因组合成技术的进步,我们现在已经可以合成原核生物(例如大肠杆菌、支原体)的基因组,甚至是合成真核生物酿酒酵母的染色体。然而,由于基因组的大小和复杂性,哺乳动物基因组合成仍然令人望而却步。
人源化小鼠模型是研究人类疾病的重要模型,然而,许多人类疾病缺乏精确的小鼠模型,这是因为在技术上很难制造广泛的遗传人源化小鼠。例如,在新冠研究中,一个重大挑战就是小鼠模型在模拟人类感染新冠后的病理生理学方面的局限性。
如果能够开发出一种更无痕、更高效的哺乳动物基因组写入(genome writing)技术,用来构建完全人源化的动物模型,这对于人类疾病的研究和药物开发帮助巨大。
2023年11月1日,纽约大学朗格尼医学中心的 Jef D. Boeke 团队(张维民博士为论文第一作者)在 Nature 期刊发表题为:Mouse genome rewriting and tailoring of three important disease loci 的研究论文。
该研究开发了一种在小鼠胚胎干细胞中大规模、高效、无痕重写基因组的方法——mSwAP-In,并展示了使用mSwAP-In迭代重写长达115kb的定制Trp53位点,以及使用人类ACE2基因替换小鼠的ACE2,从而生成人源化ACE2的小鼠模型。
值得注意的是,这一人源化ACE2小鼠模型再现了人类ACE2的表达模式和剪接方式。与现有的K18-hACE2小鼠模型相比,该模型在SARS-CoV-2病毒攻击时表现出较轻的症状,因此代表了更像人类的SARS-CoV-2动物感染模型。

基因工程小鼠疾病模型是研究人类疾病的宝贵工具,但想要在小鼠身上完全概括人类基因的复杂性是困难的。例如,如果要模拟人类COVID-19病例,就需要具有完全“人源化”ACE2的小鼠来模拟该基因的表达水平和分布模式。
目前,转基因ACE2小鼠模型已经为了解SARS、MERS和新冠病毒提供了良好的研究平台。然而,它们也存在局限性:1、缺乏人类基因组中对ACE2的调控元件,不能概括人类ACE2的时空调控;2、小鼠体内可能缺乏人类ACE2所需的剪接信号;3、转基因小鼠同时也表达内源性ACE2(鼠源),导致人和小鼠对应受体的复杂表达。
因此,迫切需要一种基因组人源化ACE2小鼠模型,来更准确地模拟新冠病毒感染。
在这项最新研究中,研究团队团队围绕着一种编辑DNA的新方法展开。区别于CRISPR基因编辑技术一次只能编辑一个或几个碱基对,这种方法选择从头开始构建DNA——在预先组装的大量基因代码中进行广泛修改,然后再递送到细胞中以代替其天然对应物。
鉴于人类基因的复杂性,研究团队首先在酵母中开发了这种“基因组写入”方法。酵母是一种单细胞真菌,与人类细胞具有许多共同特征,但更简单,更容易研究。在酵母中取得成功之后,研究团队团队将这一技术应用到哺乳动物中。
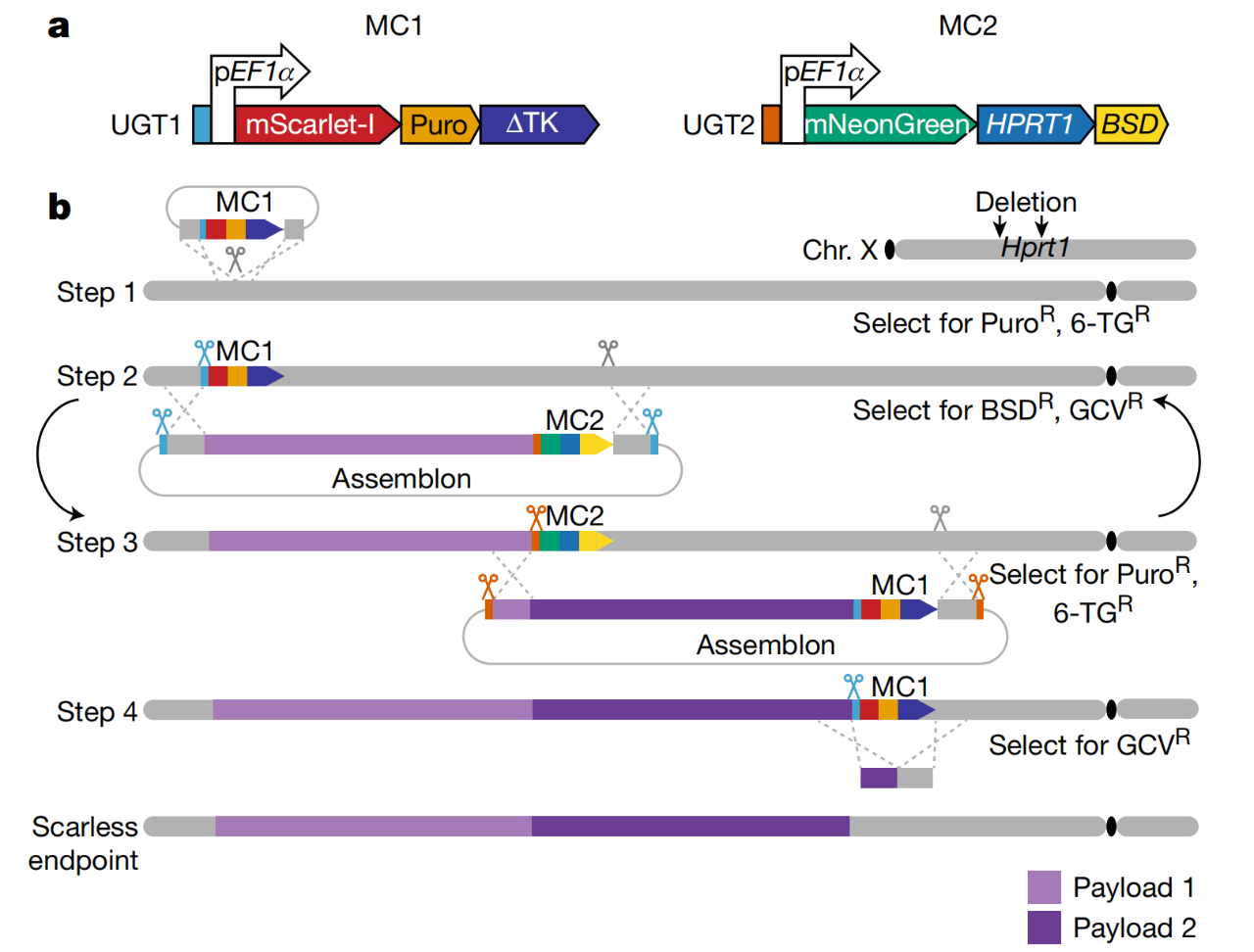
用于基因组写入的mSwAP-In策略
值得一提的是,在哺乳动物细胞中,遗传密码不仅由编码蛋白质的基因组成,还包括许多调控“开关”,这些“开关”的存在使得不同细胞类型具有极其多样化的基因表达模式,即使这些细胞具有完全相同的基因序列。
通过研究这种鲜为人知的调控基因的“暗物质”,研究团队首次能够设计出具有更接近人类ACE2基因活性水平的小鼠模型。研究团队先在酵母细胞中组装出多达20万个碱基对的DNA序列,然后使用他们的新方法——mSwAP-In将这一DNA序列递送到小鼠胚胎干细胞中。
mSwAP-In克服了过去方法的大小限制,通过用180kb的人类ACE2基因及其调控DNA“覆盖”72kb的小鼠ACE2代码,构建了人源化的COVID-19小鼠病理模型。
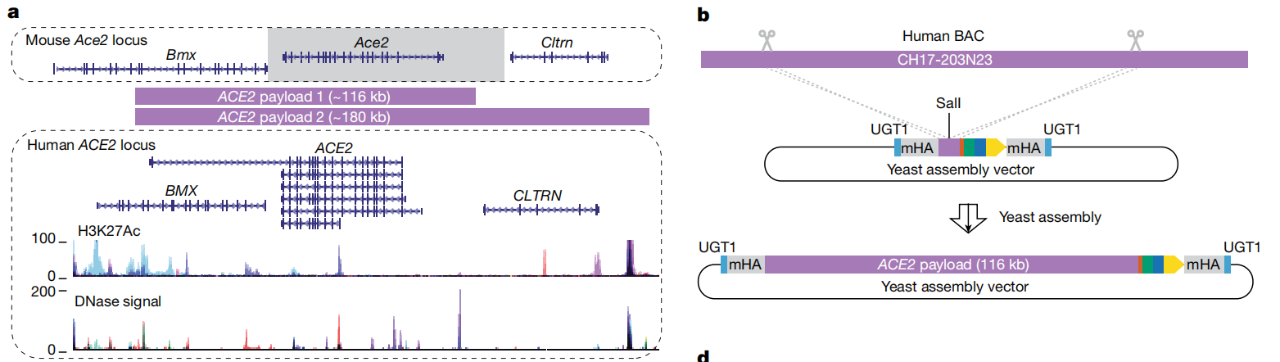
将小鼠胚胎干细胞中的ACE2完全人源化
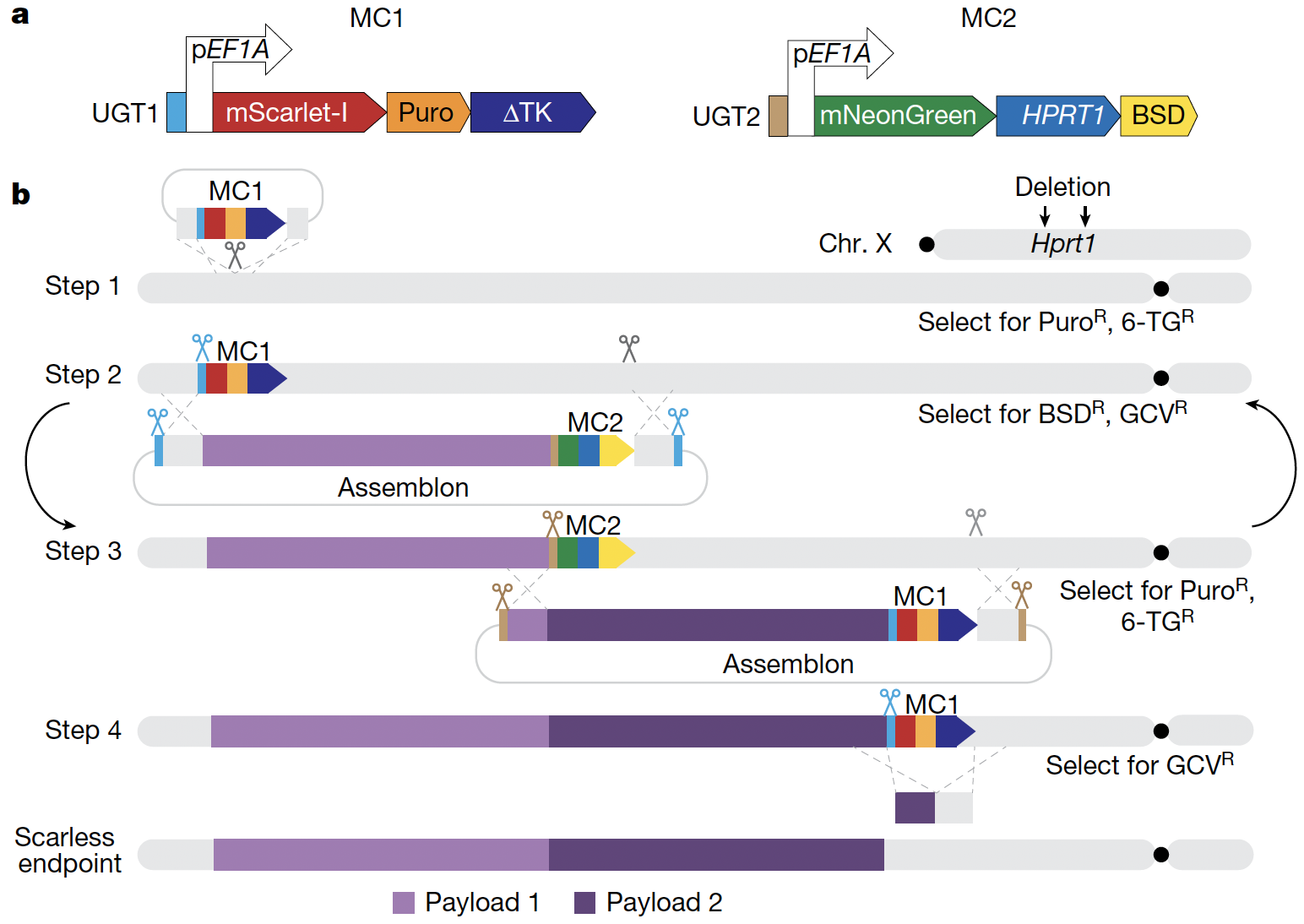
为了完成这种跨物种交换,研究团队团队在天然基因周围的DNA代码中切下一个关键位点,分步骤交换合成的对应基因,每添加一个,增加一个质量控制机制,以便只有具有合成基因的细胞存活下来。
随后,研究团队与纽约大学啮齿动物基因组工程实验室的Sang Yong Kim合作,使用一种称为“四倍体互补”的干细胞技术来制造一只活小鼠,其细胞包含覆盖的基因,从而生成了ACE2人源化小鼠模型。
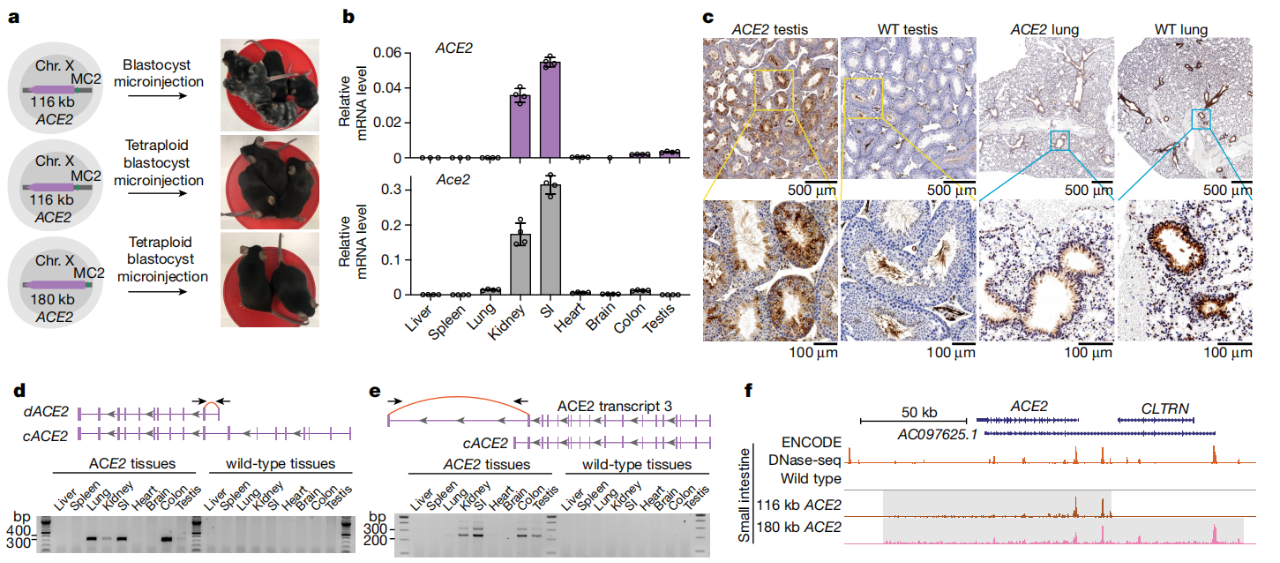
小鼠中ACE2表达的表征
进一步研究显示,该研究构建的ACE2人源化小鼠在鼻内感染时对SARS-CoV-2易感,但与K18-hACE2转基因小鼠不同,这些小鼠不会死于感染。这表明ACE2人源化小鼠是研究COVID-19更好的动物模型。
不仅如此,研究团队进一步在ACE2人源化小鼠的胚胎干细胞中用人源TMPRSS2覆盖小鼠TMPRSS2,证明了mSwAP-In的双基因的基因组写入能力,从而获得了ACE2和TMPRSS2双人源化小鼠。
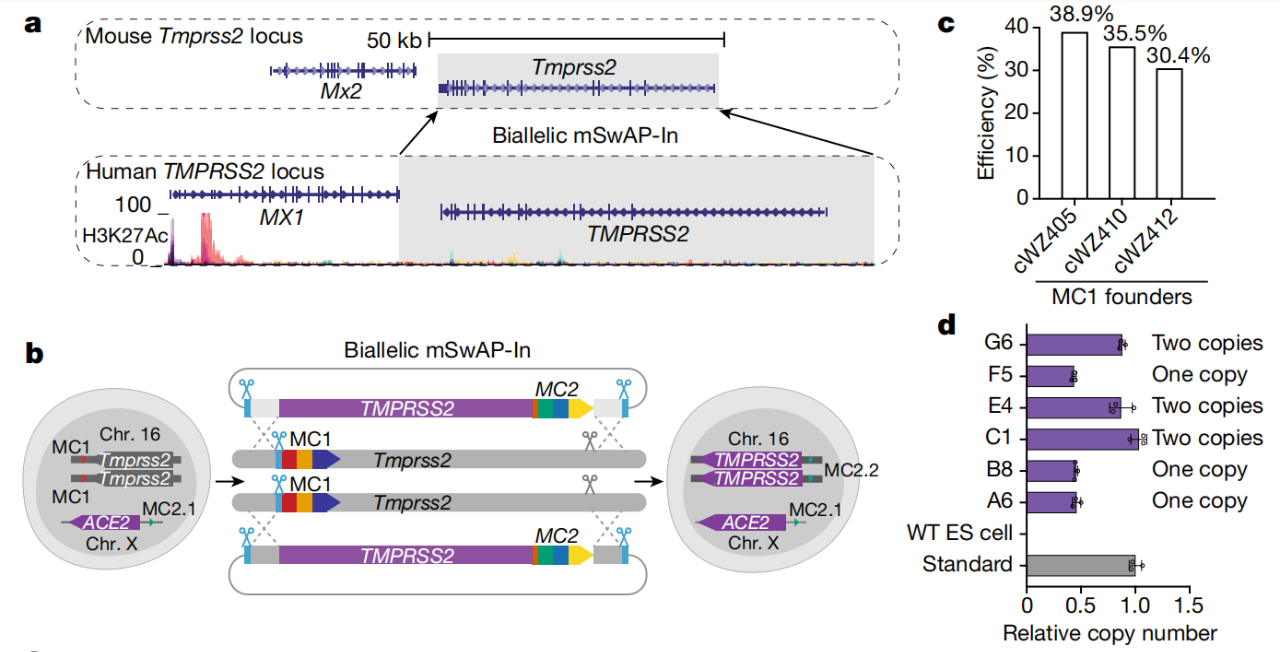
构建ACE2和TMPRSS2双人源化小鼠模型
此外,研究团队此前还设计了一种合成版本的Trp53基因(人类抑癌基因P53在小鼠中的对应基因),并将其交换到小鼠细胞中。该基因编码的蛋白质协调细胞对受损DNA的反应,甚至可以诱导细胞死亡,以防止细胞癌变。因此,当这个“基因组守护者”本身出现缺陷时,它就成了人类癌症的主要诱因。
ACE2实验中替换的是一个未改变的人类基因,而人工合成的、被替换的Trp53基因被设计成一种更稳定的形式——研究人员用包含AG的序列覆盖了关键的CG易突变位点,从而降低了Trp53基因的脆弱性,预计这种替换将使其突变率降低到原来的1/50到1/1。

论文第一作者张维民博士,张维民博士毕业于清华大学,现为纽约大学朗格尼医学中心的研究助理教授
论文第一作者张维民博士表示,团队的下一个目标是在活体动物上证明替换Trp53基因是否会导致更少的基因突变和更少的肿瘤。
用mSwAP-In重写Trp53基因
总的来说,这项发表在 Nature 的研究开发了一种全新的DNA编辑技术——mSwAP-In,该方法可用于对小鼠中的任何基因或一组基因进行人源化,从而能够快速生产动物模型,而无需通常需要耗时的育种过程。随着DNA合成成本的下降,合成和替换更大的基因组片段、甚至重写整个小鼠染色体将成为可能,从而更方便科学研究揭秘基因组的运作模式。
人类基因组庞大且复杂,DNA突变、染色体异位、染色体重复、转座子插入等基因组序列异常会引起不同类型的疾病。小鼠模型可以帮助我们解析人类疾病的致病机理,但往往由于两物种基因组在进化上的差异造成解析偏差。如何利用小鼠精确地模拟人类疾病发病过程对于治疗指导和药物开发至关重要。传统的人源化小鼠基于转基因技术,忽略了基因表达的位置和剂量效应,且删除了庞大的内含子序列,无法产生可变剪切后的多种转录单元,导致以偏概全。更重要的是由于受到基因组编辑技术的限制,人们很难将具有远程调控功能的增强子一并人源化,最终也会导致人源化基因的调控和生理条件下的偏差。
近日纽约大学医学院Jef Boeke实验室在Nature发表长文,报道小鼠基因组大片段高效编辑技术,首次实现双等位基因同时大片段编写。文章利用该技术在小鼠胚胎干细胞(mESC)中对新冠受体ACE2全基因进行人源化,并构建出在感染SARS-CoV-2后和人具有高度相似表型的小鼠模型。在随后开发双等位基因编辑技术的过程中,作者首次建立了ACE2受体和TMPRSS2细胞表面丝氨酸蛋白酶2同时大片段人源化的小鼠模型,对于研究长新冠(Long-COVID)以及快速应对突发因冠状病毒引起的疫情提供了宝贵的实验资源。更重要的是,该工作为未来基因组–编写(GP-Write)计划开辟了技术路线。

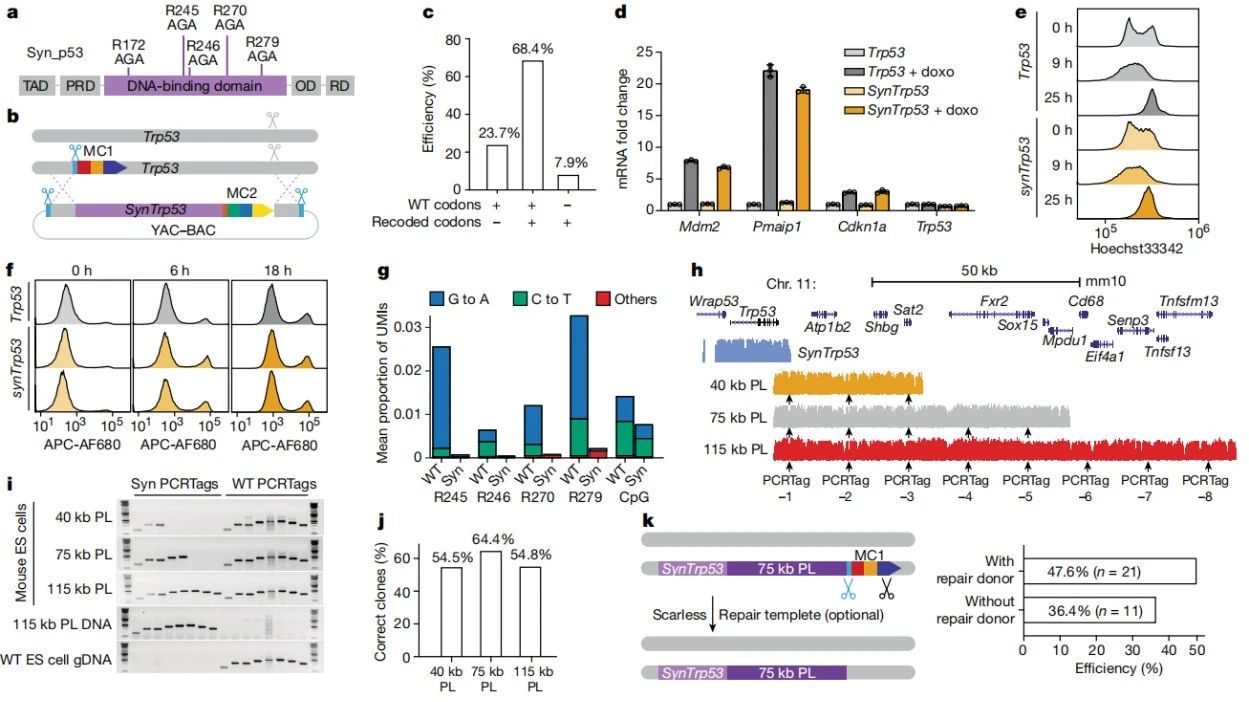
人工设计基因组序列,并最终合成具有生命特征的生命体能够帮助我们更好地理解基因组工作原理,其产物也可以在方方面面为人类生活提供便利。近二十年来,人们先后对于原核生物和单细胞真核生物的基因组实现了人工设计于合成,但是对于更庞大、更复杂的哺乳动物基因组尚没有可以被广泛应用的技术手段。而哺乳动物作为重要的实验模型,往往更多的被应用在临床研究前期的机理探究和药物筛选中。如果不能高效地编写哺乳动物基因组,我们提升健康水平的能力将大大受限。Jef Boeke实验室早在酵母基因组项目(Sc2.0 project)中就发挥了引领作用,是资深的基因组编写专家。近期他们将基因组多步迭代替换的思路应用到小鼠胚胎干细胞中,设计了哺乳动物大片段改写的方案(图一),mSwAP-In (mammalian Switching Antibiotic markers Progressively for Integration) 。作者首先以小鼠p53基因位点为例,在不改变p53蛋白序列的前提下对癌症高频突变位点(mutationhotspots) 进行重新编码,目的是降低因水解以及其他外源压力对甲基化CpG位点造成的高频突变。作者证明mSwAP-In可以在mESC中高效的改写Trp53序列。随后作者通过Amplicon-Seq证明含有重编Trp53序列的mESC在连续传代近4个月之后,其癌症高频突变位点突变率显著降低。接下来为了证明mSwAP-In可以进行多轮迭代编辑,作者对Trp53下游最长115 kb区域进行改写,通过引入水印(watermarks)序列,发现高于40%的克隆含有插入的水印序列。最终作者通过CRISPR-Cas9或PiggyBAC-excision方法将留在基因组中的筛选基因移除,实现无痕编辑。
图一,哺乳动物基因组编写策略
接下来作者尝试利用mSwAP-In对小鼠胚胎干细胞进行新冠病毒受体ACE2大片段人源化编写。为了尽量多的保有ACE2基因在内源状态下的时空调控,作者选取了大量有可能包含启动子和增强子的基因间序列以及全部内含子序列,总长度为116 kb 或 180 kb。作者利用酵母作为工具,分别将ACE2全长基因克隆到mSwAP-In递送载体上,最终精准替换了小鼠Ace2基因区域。将ACE2人源化的小鼠胚胎干细胞培育成成年小鼠之后,正如作者所预期的,人源ACE2的时空表达谱在小鼠的体内得到了很好的重现。例如野生型小鼠原本并不像人类一样在睾丸中表达ACE2蛋白,但是经过人源化后的小鼠在睾丸中高水平表达ACE2蛋白。另外,因为作者保留了所有内含子序列,人源ACE2基因在小鼠体内可以被很好地可变剪切,产生了多种人特意性转录单元,有的甚至参与机体干扰素响应过程。最后,作者通过ATAC-Seq实验证明ACE2基因区域在小鼠中也同时保留了原有的可及性。
那么具有表达水平更贴近生理水平的人源化ACE2小鼠是否可以被新冠病毒感染呢?作者和传统转基因小鼠模型K18-hACE2做了SARS-CoV-2感染实验对比,发现K18-hACE2小鼠在感染之后的6-8天之内体重剧烈下降并最终全部死亡。而本文构建的ACE2人源化小鼠全部存活,并在血液样本中检测到中和抗体(图二)。正如绝大多数人类感染新冠病毒之后能够通过体液反应恢复。
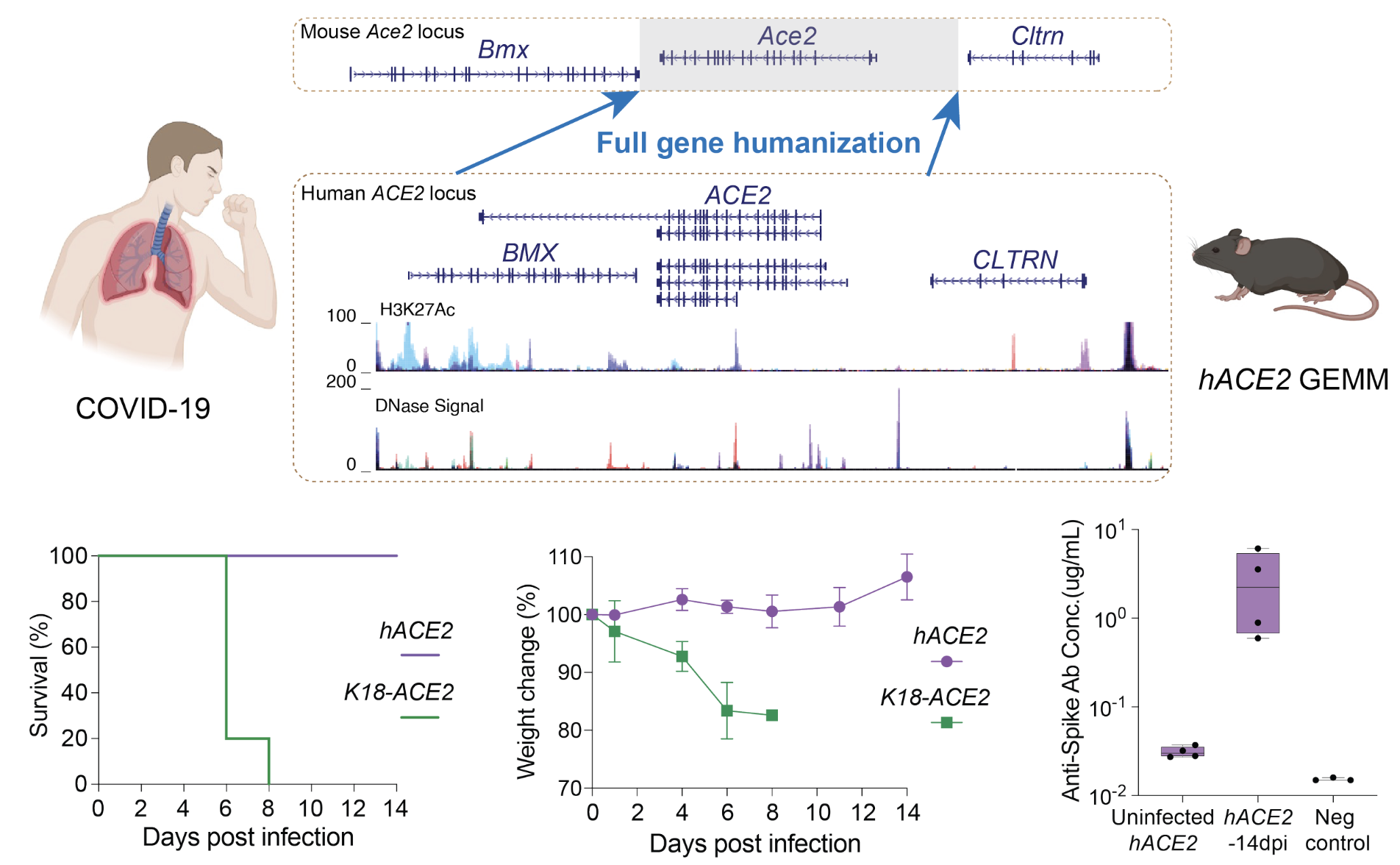
图二,ACE2全基因人源化小鼠模型的设计、构建和测试
最后,作者将大片段基因组编辑推上了一个新的高度 –在ACE2人源化的小鼠胚胎干细胞中同时对两个TMPRSS2等位基因进行编写。TMPRSS2编码细胞表面丝氨酸蛋白酶2,是介导新冠病毒入膜的另一个关键因子。在利用biallelic mSwAP-In对TMPRSS2人源化后,作者利用小鼠四倍体胚胎回补(tetraploidembryo complementation)方法培育出了ACE2, TMPRSS2双基因人源化的小鼠模型,该方法可以省略繁琐耗时的杂交过程,大大缩短了建立一个全新小鼠模型的时间。作者认为人源化ACE2受体和TMPRSS2丝氨酸蛋白酶2可以进一步提高对新冠病毒感染过程的模拟精准度,对于未来可能出现的疫情响应具有重要价值。
本文第一作者张维民于2012-2017年期间博士就读于清华大学生命科学学院,曾获清华大学研究生特等奖学金。现任职纽约大学医学院系统遗传学研究所Research Assistant Professor。博士期间在Science杂志发表封面文章报道人工合成酿酒酵母基因组编写工作。本篇Nature文章是基因组编写从单细胞真核生物向高等哺乳动物基因组编写迈进的重要一步。正如文章讨论部分中提到的,该技术不但适用于小鼠基因组人源化编写,还可应用于其他重要模式哺乳动物基因组编写,为构建多种精确的人类疾病动物模型埋下伏笔。
原文链接:https://www.nature.com/articles/s41586-023-06675-4
Nature Briefing链接:https://www.nature.com/articles/d41586-023-03079-2
近日,教育部办公厅公布了《教育部关于2022年度高等学校科学研究优秀成果奖(科学技术)奖励的决定》,自动化系张学工教授主持完成的“基因差异表达及调控的模式识别”项目和宋士吉教授主持完成的“数据或计算资源受限的高效学习理论与方法”项目,荣获2022年度教育部高等学校科学研究优秀成果奖自然科学奖一等奖;刘云浩教授主持完成的“大规模自组织物联网关键技术及应用”项目荣获技术发明奖一等奖;张毅教授主持完成的“智能车路协同与道路交通优化控制关键技术及应用”项目荣获科学技术进步奖二等奖。

基因差异表达及调控的模式识别
该项目完成人包括:张学工、汪小我、谢震、李衍达、古槿、邓柯。基因差异表达及调控的模式识别是解密生命的基础,面临超高维、极小样本、高噪声和基础原理未知等重大挑战。项目组从信息和控制视角进行了十几年持续研究取得关键突破:极小样本差异信号精准检测方法;关键调控元件精准机器学习识别方法;分子竞争调控理论与定量数学模型。该项目推动了疾病认识和科学发现、创新药进入临床试验,带动了相关学科发展。

数据或计算资源受限的高效学习理论与方法
该项目完成人包括:宋士吉、张长水、黄高、游科友、吴澄。项目针对数据或计算资源受限环境,提出了基于随机特征的半监督与无监督高效学习、跨域分布不变特征自适应学习、深度卷积网络的轻量化结构与剪枝方法等高效学习理论与方法。项目成果用于解决智能医疗诊断、空气质量监测与预报、产品表面质量检测、深海目标识别等国家重大需求问题,提升了诊断、监测、检测及识别的效果,经济和社会效益显著。

大规模自组织物联网关键技术及应用
项目完成人包括刘云浩、杨铮、马强、苗欣、刘卓、邹卫明。面向大规模自组织物联网,项目组首创了轻量级被动式网络故障诊断理论和细粒度跨协议传输方法,发明了基于本征指纹的物联网定位方法和基于无应力指纹空间的信号指纹地图自动构建机制,形成了一套构建大规模自组织物联网的理论和技术体系,所研制的产品走出了国门,有效支持了中国企业在通讯基础设施薄弱的海外地区开展工作,提高中国企业“走出去”战略的质量。

智能车路协同与道路交通优化控制关键技术及应用
该项目完成人包括:张毅、姚丹亚、裴欣、胡坚明、张佐。基于V2X的车路协同技术是当今国际智能交通领域的前沿技术和必然发展趋势,是提高效率、保证安全、降低排放的有效手段,从根本上改变了传统道路交通的发展模式;同时也将单车智能的自动驾驶升级到车路协同的智能驾驶,开创了我国车路协同自动驾驶新的发展战略。项目主要创新点包括:发明了基于车路协同的中国智能交通系统体系框架并制定了我国智能车路协同数据通信国家标准;攻克了面向智能管控新业务的车路协同关键支撑技术;提出了基于车路协同的道路交通优化控制关键技术;填补了智能车路协同集成与优化控制关键技术应用的空白。
高等学校科学研究优秀成果奖(科学技术)是由教育部设立的用以鼓励在推动科学技术进步中做出突出贡献的高等学校的教师、科技工作者和科研组织的奖项, 是仅次于国家自然科学奖、国家技术发明奖和国家科技进步奖三大奖的重要科技奖项。其评审面向全国高等院校,由全国同行专家审议选定,竞争激烈。
6月11日-15日,2023第15届国际代谢工程大会(15th Metabolic Engineering Conference,简称ME15)在新加坡隆重举办。该大会由代谢工程领域的国际权威学术组织国际代谢工程学会(International Metabolic Engineering Society,简称IMES)主办。
本届大会围绕“代谢工程中的大数据与人工智能”“代谢工程与下一代医药”“合成生物学与代谢工程”“生物铸造厂:代谢工程中的自动化”“代谢工程与生物制造:从实验室到工业化”“代谢工程与下一代底盘”等议题展开,多方位展示了代谢工程领域近两年的最新科技成果及未来研究热点。
大会汇集了全球代谢工程领域的顶尖科学家,美国工程院外籍院士、韩国国家科学与工程院院士,现任韩国科学技术院长Sang Yup Lee,国际合成生物学产业化先驱、美国国家工程院院士、劳伦斯伯克利国家实验室、美国加州大学伯克利分校合成生物学工程研究中心主任Jay Keasling,荷兰代尔夫特理工大学工业微生物学教授兼生物技术系主任、荷兰科学界最高奖项史蒂文奖获得者Jack Pronk等,还有来自美国、德国、英国、中国、日本、韩国、新加坡等28多个国家的671位代谢工程领域专家学者及相关产业届代表参会,规模为历届之最。

大会现场图
6月14日会议期间,举行了重要的2023年度国际代谢工程奖(IMES Award)颁奖仪式,清华大学生命科学学院、化工系教授、合成与系统生物学中心主任陈国强为本届该奖项唯一获奖者,也是首个获得该项荣誉的中国大陆地区学者。同时,陈国强教授在ME15大会上作题为“Next Generation Industrial Biotechnology Based on Synthetic Biology of Halomonas spp.”的特邀报告,分享其在代谢工程领域的最新研究成果。

陈国强教授获国际代谢工程奖(IMES Award)
陈国强教授为全球顶尖合成生物学专家,代谢工程领域的领军型人物,清华大学教授、合成与系统生物学中心主任,国家973“合成生物学”项目以及国家重大专项项目的首席科学家,也是中国大陆首位国际代谢工程奖获得者。
陈国强教授同时在全球合成生物学顶级期刊 Synthetic and Systems Biotechnology(《合成和系统生物技术》,简称SSB)担任共同主编,并在国际学术期刊如Chemical Reviews 、Chemical Society Reviews等上共发表微生物技术和生物材料相关论文400多篇,Web of Science引用两万五千多次,H指数78。获得授权专利50多项,以及50个公开专利。
此次陈国强教授获得的国际代谢工程奖,由国际代谢工程学会发起,每两年授予一位在代谢工程领域做出卓越贡献的研究人员,并在其和美国化学工程师协会(AIChE)共同举办的国际代谢工程大会上颁奖。自2000年该奖项正式颁发以来,获奖者均来自海外,包括生物化学工程先驱James E.Bailey、合成生物学领域权威Jay Keasling,以及多位美国工程院院士等顶尖科学家,陈国强教授是首位获得该奖项的中国学者。
在颁奖典礼上,国际代谢工程奖评选委员会主席、韩国科学院化学与生物分子工程系杰出教授、代谢与生物分子工程国家实验室主任Sang Yup Lee院士介绍了该奖项的评审情况,他表示:“陈国强教授团队最初采用了基于传统微生物底盘的工业生物技术(CIB),如使用大肠杆菌等作为底盘,然而在放大过程中存在染菌的问题。这促使他对嗜盐菌Halomonas进行开发,避免了染菌问题,并且实现连续开放、无需灭菌的发酵过程,这一技术已被广泛采用并应用于化学品的开放生产。因此组委会决定将2023国际代谢工程奖颁发给陈国强教授,以表彰他通过开发嗜盐菌和‘下一代工业生物技术’对代谢工程领域做出的杰出贡献。恭喜陈国强教授!”

Sang Yup Lee院士(左)与陈国强教授(右)
资料显示,相较于传统化工制造,以合成生物学为核心的生物制造通常以可再生生物资源为原料,可摆脱对石油资源的依赖,降低能耗,大幅减少污染物生成,具有高效、绿色、可持续的优势特性。但由于微生物原有的生物系统限制,工业化生产过程中往往会遇到许多技术瓶颈。陈国强教授长期从事“生物合成PHA材料及其下一代工业生物技术(Next Generation Industrial Biotechnology, 简称NGIB)”的研究,利用合成生物学和代谢工程学方法,构建了更加符合规模化生产的工程菌株和技术体系,实现了生物制造产业关键核心技术的不断突破。
2005年,陈国强教授团队在位于世界最酷热、最干燥的地区之一、盐浓度达200克/升的中国新疆艾丁湖筛选出适应力最强的工业微生物菌株——嗜盐菌Halomonas,通过基因编辑、代谢网络调控等技术,重新构建了嗜盐菌的代谢网络,改造出适应能力更强、生长速度更快的菌株,并开发出适用于该底盘菌株的NGIB体系,覆盖了理论、模型、分子操作、实验室培养技术、中试技术及工业生产技术,以及部分产品的应用等。目前在该技术体系下,菌株已连续超过20代迭代。
相比于利用传统微生物底盘的工业生物技术(CIB),如大肠杆菌等,基于嗜盐菌的NGIB无需灭菌,生产过程可以开放和连续化,降低了能源和淡水消耗,并且降低了生物制造的复杂性和设备的制造成本,使生物制造产品竞争性得到增强。陈国强教授团队预测,NGIB能够将传统生物制造的能耗降低50%以上,并大幅提高生产效率。
对于获得2023年度国际代谢工程奖,陈国强教授表示:“我很荣幸能够得到这一奖项认可,也很激动能够再次与大家相聚在代谢工程领域的学术盛会。非常感谢国际学术界同仁一直以来的交流与帮助,是他们的杰出成就推动了代谢工程领域的发展,也非常感谢我的团队、学生为‘下一代工业生物技术’所作出的贡献。在各位学术同仁的努力下,代谢工程和合成生物学正在对更多领域产生积极的影响,我相信这些技术未来将扮演更加重要的角色,也期待能够为推动学术界、产业界发展做出更大的贡献。”
《人民日报》曾评价:“在合成生物学和‘下一代工业生物技术’制造PHA生物塑料的道路上,陈国强团队使我国处于世界领先的水平。”目前,陈国强教授开发的技术已经在合成生物学产业中得以应用,国内已有多家企业如微构工场、微琪生物、安琪酵母等已应用最新的NGIB技术,进行规模化生物制造。可以说,受到国际认可,并实现了产业化落地的NGIB体系,前景无限可期。
原文转载自中国科技新闻网:https://www.zghy.org.cn/item/689912575852269568
3月24日,国际代谢工程学会(IMES)在其官方平台(https://www.aiche.org/community/bio/george-guo-qiang-chen)重磅宣布,将2023年度国际代谢工程奖(IMES Award)授予中国学者,清华大学生命科学学院教授、合成与系统生物学中心主任陈国强,以表彰他通过开发利用嗜盐菌和“下一代工业生物技术(NGIB)”对全球代谢工程领域所做出的卓越贡献。
国际代谢工程奖由代谢工程领域的国际权威学术组织国际代谢工程学会(IMES, International Metabolic Engineering Society)发起,每两年授予一位在代谢工程领域做出卓越贡献的研究人员,并在其和美国化学工程师协会(AIChE)共同举办的国际代谢工程大会(Metabolic Engineering Conference)上颁奖。

国际代谢工程学会(IMES)致力于促进和推进代谢工程,将其作为材料、药品、食品配料、化学品和燃料生物生产的一门辅助科学。2023年的第15届国际代谢工程大会将于6月11日至15日在新加坡举行,届时陈国强教授也将出席大会,并向全球学者介绍下一代工业生物技术(NGIB)在生物制造领域的最新进展。
IMES在官方网站上的展示
陈国强教授成为首位获得IMES Award的中国科学家
自2000年国际代谢工程奖正式颁发至今,陈国强教授成为了首位获得该奖项的中国科学家。此前获得该奖项的科学家包括生物化学工程先驱James E.Bailey、合成生物学领域权威Jay Keasling,以及多位美国工程院院士。
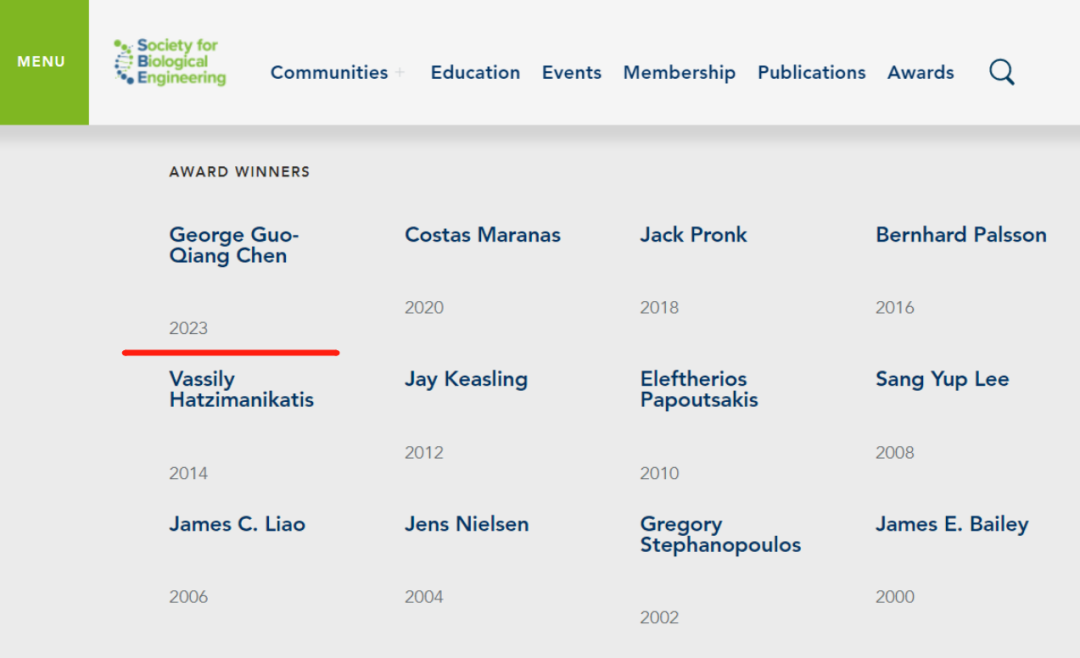
历届IMES Award获奖者名单
国际代谢工程奖评选委员会主席Sang Yup Lee教授表示:“陈国强教授一直致力用合成生物学的方式对嗜盐菌进行研究开发,以进行生产多种PHA和其他小分子化合物,对代谢工程领域做出了突出的贡献。目前,工程极端微生物和‘下一代工业生物技术’的概念已被广泛采用并应用于化合物的开放生产,陈国强教授获得国际代谢工程奖实至名归。”
NGIB将为生物制造领域带来颠覆性变革
2005年,陈国强教授和科研团队在位于世界上最酷热、最干燥的地区之一的新疆艾丁湖筛选出适应力最强的工业微生物菌株——嗜盐菌Halomonas,利用合成生物学和代谢工程学方法,通过DBTL(设计-构建-测试-学习)体系重新编辑嗜盐菌的基因,改造出适应能力更强、生长速度更快的菌株,成功开发了NGIB,覆盖了理论、模型、分子操作、实验室培养技术、中试技术及工业生产技术,以及部分产品的应用等。
相比于利用传统微生物底盘的工业生物技术(CIB),基于嗜盐菌的NGIB无需灭菌,生产过程可以开放和连续化,降低了能源和淡水消耗,并且降低了生物制造的复杂性和设备的制造成本,使生物制造产品竞争性得到增强。陈国强教授团队预测,NGIB能够将传统生物制造的能耗降低50%以上,并大幅提高生产效率。

合成生物学企业微构工场、微琪生物、安琪酵母、
麦得发、中粮生化等已应用最新的NGIB技术,
进行规模化生物制造
陈国强教授前期利用传统的微生物底盘对PHA材料进行过大规模的生产,发现了染菌问题很难解决,进而对嗜盐菌进行多年的代谢工程以及合成生物学改造,以令其更适用于大规模生物制造,包括重构Halomonas底盘细胞底物代谢途径,使其能利用多种底物,启动子工程和血红蛋白的配置以及新型反转搅拌发酵罐,使利用氧气效率大幅提升;重编群体效应代谢通路、形态学工程,改变细菌的生长方式、改造嗜盐菌的表面电荷,使细菌自凝絮,有利于连续发酵和菌分离;开放式的发酵减少了工艺流程和设备的复杂性等,目前该菌株已连续超过20代迭代。
合成生物学改造的嗜盐菌体系,也成功实现了各种生物及化学品的成功生产,括PHB、P34HB、P34HBHV、PHBHHX、PHB5HV、P3HP3HB等多种PHA材料。此外,还可用于生产戊二胺、四氢密呢、肌醇、氨基酸、3-轻基丙酸等产品。目前已获得的各种PHA材料正在开发用于医疗组织工程、功能材料、薄膜、纤维以及3D打印等领域,PHA材料的降解产物3-羟基丁酸及其衍生物也可用于研究治疗骨质疏松和老年痴呆等。

利用NGIB技术生产的PHA颗粒
不仅如此,在非粮碳源的利用上,陈国强教授团队也屡屡斩获突破。经过改造后的嗜盐细菌,可以利用秸秆糖、餐厨废料、废糖蜜、废工业乙酸等原料来合成PHA材料,为农业废弃秸秆的高值利用储备了先进技术,实现了非粮碳源生产。
目前,陈国强教授团队开发的技术已经在合成生物学产业中得到了应用,《人民日报》曾评价:“在合成生物学和‘下一代工业生物技术’制造PHA生物塑料的道路上,陈国强团队使我国处于世界领先的水平。”
个人简介
陈国强,1985年本科毕业于华南理工大学,1989年获得奥地利格拉茨(Graz)工业大学博士学位,1990至1994年在英国诺丁汉(Nottingham)大学和加拿大阿尔伯达(Alberta)大学做博士后研究,1994年回国入职清华大学生物系,现为清华大学生命科学学院教授、清华大学合成与系统生物学中心主任。

陈国强教授长期从事“生物合成PHA材料及其下一代工业生物技术”的研究,他在包括Chemical Reviews、Chemical Society Reviews、Science、Nature Communications等在内的多个国际学术期刊上发表微生物技术和生物材料相关论文380多篇,Web of Sciences记录论文被引用25000多次,已经获得50多项授权专利,以及50个公开专利。
陈国强教授以往获得的荣誉还包括:全国先进科技工作者(2016)、候德傍化工创新奖(2015)、首届闵恩泽能源化工杰出贡献奖(2013)、谈家祯生命科学创新奖(2011)、第八届中国青年科技奖(2003)、纽伦堡国际发明奖(2003)、茅以升科技奖(2003)、国家发明二等奖(排名第一)(2002)等。他还曾连续6年获得清华大学学生“良师益友”的光荣称号,进入清华大学“良师益友”名人堂,连续9年获得清华大学高论文他引的“梅贻琦奖”,连续7年获得Elsevier出版社生化与分子生物学高引用作者。此外,他还担任了Synthetic and Systems Biotechnology(SSB)的共同主编及多家国际、国内学术期刊的副主编职务。
原文转自:清华生命学院
清华大学生命科学学院教授陈国强受邀清华大学智能产业研究院(AIR),作题为《嗜盐菌合成生物学和“下一代工业生物技术”》的报告。

【讲者介绍】
陈国强教授是清华大学生命科学学院长聘教授,博士生导师,教育部长江学者特聘教授,清华合成与系统生物学中心主任;曾担任973计划、国家重点研发计划“合成生物学”专项首席科学家;长期从事“生物合成PHA材料及其下一代工业生物技术”的研究。
陈国强教授在国际学术期刊如Chemical Reviews, Chemical Society Reviews, Science, Advanced Materials, Advanced Science, Nucleic Acids Research, Nature Communications, Trends in Biotechnology, Biometaerials, Metabolic Engineering, Current Opinions in Biotechnology等上共发表微生物技术和生物材料相关论文350多篇,Web of Science纪录论文被引用两万两千多次(H指数为72)[Google Scholar引用六万多次,H指数100]。获得授权专利50多项,50个公开专利。他开发的技术已经在数家公司用于大规模生产微生物塑料聚羟基脂肪酸酯PHA。
【预约参会】
陈国强教授于3月20号(周一)下午14:00的讲座,欢迎大家预约线下参会,与讲师现场互动:
扫码线下预约参会

原文从“清华大学智能产业研究院”公众号转载,感兴趣的老师可以搜索并关注。
教育工作经历
1998-2002 武汉大学化学与分子科学学院 学士
2002-2008 中国科学院生物物理研究所 博士
2009-2015 美国国立卫生研究院国立癌症研究所 博士后
2015-至今 清华大学生命科学学院 研究员、博导
研究方向:
课题组主要聚焦于RNA的整合结构生物学研究。近年来,我们发展了长链RNA的位点特异性标记技术(Nat.Commun. 2021,PNAS 2020,Chem. Sci. 2020),发展了长链RNA及其蛋白质复合物高级结构研究的整合方案;另一方面,我们整合运用小角X射线/中子散射、电子顺磁共振/核磁共振、X射线晶体学、单分子荧光共振能量转移/单分子生物纳米孔技术以及计算模拟,研究了黄病毒来源的功能RNA的高级结构、动态特性、相互作用与功能的关系(Biophys.J. 2022,Nat.Commun. 2021,Nat. Commun. 2020, EMBO Rep 2019)。此外,课题组在致病细菌功能性淀粉样蛋白的结构与形成机制方面也进行了有益探索(EMBO J. 2021,JBC 2018)。课题组将继续开发RNA结构研究的新方法,并应用相关方法开展与重大疾病和关键生命过程密切相关的非编码RNA的结构生物学研究,开展基于结构的mRNA分子的理性设计与优化研究。
发表论文
联系方式:
Email: 该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
Tel: +86-10-62771071
王海峰
清华大学 生命科学学院
博士,助理教授
教育经历
2000-2004 中国农业大学 本科
2004-2007 清华大学 硕士
2007-2013 加州大学戴维斯分校 博士
2013-2014 加州大学戴维斯分校 博士后
2014-2021 斯坦福大学 博士后
2021-现在 清华大学生命科学学院 助理教授
2021-现在 清华大学生命科学联合中心 研究员
研究方向:
本实验室将在哺乳动物细胞及模式生物中,开发利用CRISPR技术研究细胞发育生物学和疾病相关机理,并应用于精准医学诊断治疗。具体研究方向包括:
(1)开发CRISPR合成生物学工具从多维度研究基因组和转录组的调控机理;
(2)利用新型工具探求疾病发育过程中基因组和转录组的时空变化和基因调控的机理;
(3)精准医学:针对疾病相关的基因组或转录组异常,开发相应的诊断和靶向治疗方法。
代表性论文:
1,Wang H, Han M, Qi LS, Engineering 3D genome organization, Nature Review Genetics, 2021 Feb; PMID: 33558716;
2,Wang H, Nakamura M, Abbott TR, Zhao D, Luo K, Yu C, Nguyen CM, Lo A, Daley PT, La Russa M, Liu Y, Qi LS, CRISPR-mediated live imaging of genome editing and transcription. Science, 2019 Sep; Vol. 365, Issue 6459, pp. 1301-1305.
3,Wang H, Xu X, Nguyen CM, Liu Y, Gao Y, Lin X, Daley PT, Kipniss NH, La Russa M, Qi LS, CRISPR-Mediated Programmable 3D Genome Positioning and Nuclear Organization. Cell, 2018 Oct; 175, 1405-1417 e1414.
4,Wang H, La Russa M, Qi LS, CRISPR-Cas9 in genome editing and beyond. The Annual Review of Biochemistry, 2016 Aug; Vol. 85: 227-264.
5,Wang H, Brust-Mascher I and Scholey JM, The microtubule cross-linker Feo controls the midzone stability, motor composition, and elongation of the anaphase B spindle in Drosophila embryos. Molecular Biology of the Cell. 2015 Apr 15; 26(8):1452-62. PMID: 25694445. Selected as the Cover paper.
6,Wang H, Brust-Mascher I and Scholey JM, Sliding filaments and mitotic spindle organization, Nature Cell Biology, 2014 Aug; 16(8):737-739. PMID: 25082196.
7,Wang H, Brust-Mascher I, Civelekoglu-Scholey G and Scholey JM, Patronin mediates a switch from kinesin-13-dependent poleward flux to anaphase B spindle elongation, Journal of Cell Biology, 2013 Oct; 203(1):35-46. PMCID: PMC3798244.
联系方式:
邮箱:该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
2021年5月11日,聚焦合成生物学领域的Synbio Hive 创新孵化平台首场路演在清华大学生命学院顺利举行。该平台的主题是“Engineering Biology, Engineering Tomorrow”,致力于打造成为中国本土的合成生物领域顶级平台。路演吸引了中心多位老师和同学、产业和资本界代表等多位业内人士关注并到场交流。
据介绍,Synbio Hive平台旗下包括创新大赛、创业大赛、企业孵化、学术峰会、产业峰会等不同板块。平台将以大赛作为依托,希望打通中国合成生物领域的产、学、研、创、投通道,真正形成中国合成生物的创新落地生态闭环,让合成生物领域的创新想法有落地的支撑和可能,以产业需求驱动源头创新。
本场路演由合成与系统生物学中心主任陈国强教授主持并致开幕词。陈国强教授开场先简要介绍了Synbio Hive成立的初衷,并希望不断集思广益改善平台赛事的举办形式和发展;随后生命学院副院长刘栋教授从广为熟知的iGEM赛事出发,结合中国特色,提出打造中国独有的合成生物学赛事平台的重要性,并当场表示会对Synbio Hive的发展提供强力的支持和帮助。
在介绍了Synbio Hive的具体运营模式之后,包括于慧敏教授、李春教授、张翀副教授以及张数一副教授在内的多位中心老师对该赛事平台进行了点评,并提出较多宝贵而又富有建设性的意见和建议,并希望能不断推动该平台的又好又快发展。以中石化资本、诺维信为代表的资本和产业界同仁也提出了较为针对性的研究成果到产业落地的意见,从而不断改善Synbio Hive平台的建设和发展。

The provision of new antibiotics to combat antimicrobial resistance (AMR) and to treat neglected tropical diseases (NTD), which are problematic in the developing world, is a major global challenge. Most of the of antibiotics in clinical use today are derived from microbial secondary metabolites (natural products). In addition to essential antibacterial, antifungal and antiparasitic agents, microbial natural products have been developed as antitumour, antiviral, immunosuppressive agents and as cholesterol lowering agents. In many cases the natural products are produced in very small quantities by microbial species that are difficult to cultivate or genetically manipulate. In addition to this, genome sequencing reveals many bacterial strains possess cryptic biosynthetic gene clusters (BGC) that appear to be silent when cultivated in the laboratory. These strains possess the machinery to produce bioactive natural products, but the biosynthetic genes are not expressed in the native hosts. One way to increase production levels, and also to activate expression of cryptic/unproductive BGC, is to transfer the BGC into heterologous host strains which are more genetically tractable and easier to cultivate. Unfortunately, there are a limited number of well characterised hosts available and some of these are not necessarily ideal for production of certain natural product scaffolds. For example, Escherichia coli and yeast have been widely used, but these strains are not known to produce large numbers of secondary metabolites and production of certain types of natural products can be problematic, particularly those generated by nonribosomal peptide synthetase (NRPS), polyketide synthase (PKS) and hybrid NRPS-PKS assembly lines. In this project we will exploit recent advances in synthetic biology to develop new genetic tools for heterologous expression and manipulation of microbial BGC in new Halomonas host strains to produce antibiotics and other important bioactive natural products. Halomonas are halophiles (tolerate high salinity) and can be easily cultivated under nonsterile conditions. In light of this, Halomonas sp. have emerged as a useful host for production of low molecular weight metabolites and also biopolymers. In this project we aim to develop Halomonas further as heterologous host for production of microbial NRPS, PKS and hybrid NRPS-PKS secondary metabolites, which provide many valuable antibiotics and other important therapeutic agents.
The project will involve a collaboration between the Micklefield lab at the Manchester Institute of Biotechnology (MIB) and George Chen, Director of the Center for Synthetic and Systems Biology (CSSB) at Tsinghua University in Beijing. The student will spend two years in Manchester and two years in Beijing. Training in Manchester will cover natural products chemistry, protein engineering, directed evolution, enzyme characterisation and enzyme assays. In Singapore the student will develop further skills in synthetic biology, molecular biology and microbiology, including manipulation of Halomonas bacteria. Candidates are not expected to have expertise in these areas at the outset; above all, scientific curiosity and a desire to work in a multidisciplinary environment are most important. Candidates with a degree in Chemistry, Biochemistry or Biological Sciences and an interest in enzyme catalysis, biosynthesis (natural products), microbiology, synthetic biology or a related science are encouraged to apply.
Entry Requirements
Students must meet the entry requirements of both universities, and will be registered at both institutions in the first year of study.
Applicants should hold (or be about to obtain) a minimum 2:1 bachelor's degree (or overseas equivalent), in a relevant discipline, plus a master's degree or extensive research experience. Applicants can be internal or external to The University of Manchester. For applicants whose first language is not English, we require a minimum IELTS score of 6.5.
For more information see:
http://www.manchester.ac.uk/research/Jason.micklefield/
http://www.micklefieldlab.chemistry.manchester.ac.uk
http://life.tsinghua.edu.cn/lifeen/info/1034/1088.htm
synbiodual