

卢磊
助理教授,博士生导师
清华大学,药学院
研究经历
威斯康星大学-麦迪逊分校,美国 2014-2020 博士(药学与化学)
佐治亚理工大学, 美国 2019-2021硕士(计算机科学)
武汉大学 2012-2014硕士(药物工程)
武汉大学 2008-2012本科(药学)
研究方向
研究背景多元化, 在蛋白质从头设计、蛋白质组学、计算生物学及化学生物学等领域有多项研究成果, 发表于 Science、 Nature Methods 等杂志。未来的研究集中于使用糖蛋白质组学和蛋白设计的方法发展新型疾病监测与治疗的技术。
代表论文
1. Lu L, Gou X, Tan S, Man S, Yang H, Zhong X, Gazgalis D, Valdiviezo J, Jo H, Wu Y, Diolaiti M, Ashworth A, Polizzi N, & DeGrado W. (2024) De novo design of drug-binding proteins with predictable binding energy and specificity. Science, 384,106-112.
2. Lu L, Scalf M, Shortreed M, Smith L. (2021) Mesh fragmentation improves dissociation
efficiency in top-down proteomics. Journal of the American Society for Mass Spectrometry, 23;32(6):1319-25.
3. Lu L*, Riley N*, Shortreed M, Bertozzi C & Smith L (2020). O-Pair Search with MetaMorpheus for O-glycopeptide Characterization. Nature Methods, 17, 1133-1138.
4. Zhong X, Yu Q, Ma F, Frost D, Lu L, Chen Z, Zetterberg H, Carlsson C, Okonkwo O & Li L. (2019). HOTMAQ: A Multiplexed Absolute Quantification Method for Targeted Proteomics. Analytical Chemistry, 91(3), 2112-2119.
5. Lu L, Millikin R, Solntsev S, Rolfs Z, Scalf M, Shortreed M, & Smith L. (2018). Identification of MS-cleavable and noncleavable chemically cross-linked peptides with MetaMorpheus. Journal of Proteome Research, 17(7), 2370-2376.
6. Li B, Li H, Lu L, & Jiang J. (2017). Structures of human O-GlcNAcase and its complexes reveal a new substrate recognition mode. Nature Structural & Molecular Biology, 24(4), 362.
7. Hu C, Worth M, Fan D, Li B, Li H, Lu L, Zhong X, Lin Z, Wei L, Ge Y and Li L, Jiang J.(2017). Electrophilic probes for deciphering substrate recognition by O-GlcNAc transferase. Nature Chemical Biology, 13(12), 1267.
8. Lu L, Fan D, Hu, C, Worth M, Ma Z, & Jiang J. (2016). Distributive O-GlcNAcylation on the highly repetitive C-terminal domain of RNA polymerase II. Biochemistry, 55(7), 1149-1158.
9. Zhu F, Lu L, Fu S, Zhong X, Hu M, Deng Z, & Liu T. (2015). Targeted engineering and scale up of lycopene overproduction in Escherichia coli. Process Biochemistry, 50(3), 341-346.
10. Liu Q, Wu K, Cheng Y, Lu L, Xiao E, Zhang Y, Deng Z and Liu T. (2015). Engineering an iterative polyketide pathway in Escherichia coli results in single-form alkene and alkane overproduction. Metabolic Engineering, 28, 82-90.
11. Liu, R, Zhu F, Lu L, Fu A, Lu J, Deng Z, & Liu T. (2014). Metabolic engineering of fatty acylACP reductase-dependent pathway to improve fatty alcohol production in Escherichia coli. Metabolic Engineering, 22, 10-21.
12. Zhu F, Zhong X, Hu M, Lu L, Deng Z, & Liu T. (2014). In vitro reconstitution of mevalonate pathway and targeted engineering of farnesene overproduction in Escherichia coli. Biotechnology and Bioengineering, 111(7), 1396-1405.
联系方式
邮箱: 该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
龚海鹏
清华大学 生命学院
副教授,博士生导师
教育工作经历
1993-1997 清华大学生物科学与技术系学士
1997-2000 清华大学生物科学与技术系硕士
2000-2006 约翰霍普金斯大学分子生物物理学博士
2006-2007 约翰霍普金斯大学分子生物物理学博士后
2007-2009 芝加哥大学生物化学和分子生物学博士后
2009-2014 清华大学生命学院助理教授
2015-至今 清华大学生命学院副教授
研究方向
代表性论文
Email: 该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
Website: http://structpred.life.tsinghua.edu.cn
秦为
助理教授,博士生导师
清华大学,药学院
个人简介
秦为博士2014年本科毕业于北京理工大学生命学院,2019年毕业于北京大学前沿交叉学院,获博士学位,师从王初教授和陈兴教授。2019年到2023年在斯坦福大学从事博士后研究工作,合作导师为Alice Y. Ting教授。近年来,以第一或通讯作者(含共同)在Cell, Nat Methods, Nat Chem Biol, Nat Commun, JACS, Angew Chem Int Ed, ACS Cent Sci等期刊发表论文多篇。 秦为博士获得2023年麻省理工科技评论中国区 35 岁以下 35 人、国家级海外人才、北京市杰青、 拜耳研究员奖等荣誉,担任国家重点研发计划青年项目首席科学家,主持国家自然科学基金面上项目、重大研究计划培育项目、北京分子科学国家研究中心开放课题等科研项目。担任中国生物工程学会系统生物医学专业委员会委员。担任Nat Biotechnol, Nat Methods, Nat Chem Biol, Nat Commun等多个期刊审稿人。
研究方向
秦为课题组长期致力于发展功能驱动的,具有时空动态分辨率的化学生物学工具和蛋白质组学技术,探索细胞间通讯,细胞器和生物分子互作,翻译后修饰和等重要生物学问题。具体研究大方向如下:
代表论文
联系方式
邮箱: 该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
课题组主页: thu-qin-lab.org
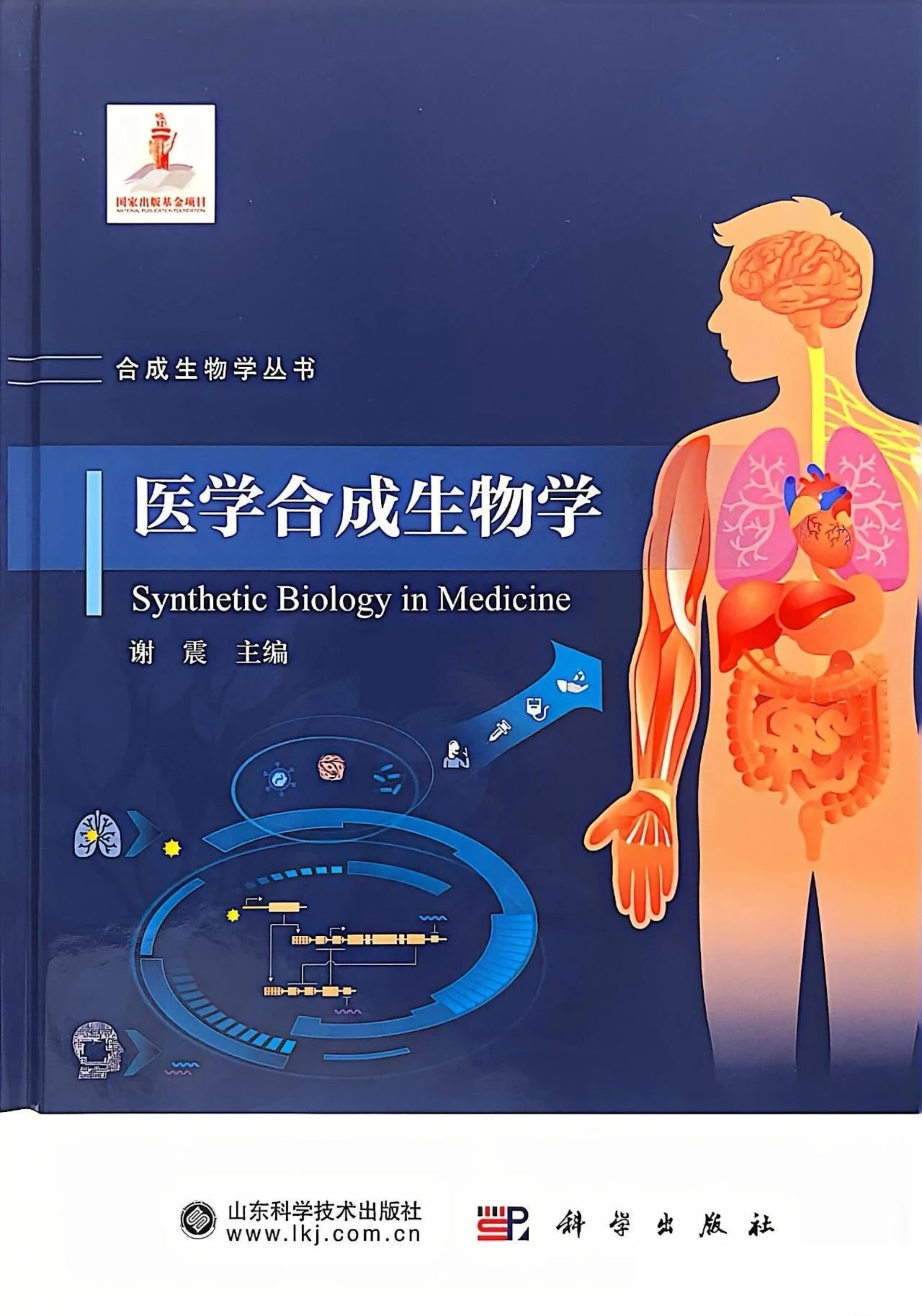
近日,金宇保灵生物药品有限公司、北京合生基因科技有限公司正式收到中国农业农村部颁发的猫传染性腹膜炎(FIP)mRNA疫苗兽用生物制品临床试验批件(批件号:2025062)。这一成果不仅标志着中国在宠物RNA原创药物研发领域的全球领先地位,更凸显了清华大学谢震教授从自动化工程跨界生物医学的重要贡献,他领导的研究团队建立了较为完整的宠物RNA药物研发体系,为“猫界绝症”带来曙光。
此次临床试验件的获得,得益于清华大学谢震教授团队与两家生物科技公司的紧密协作。据北京合生基因科技有限公司刘淦博士介绍,“在公司首席科学官谢震教授的指导下,我们组建了一支跨学科研发团队,团队紧密协作,充分融合生物技术(BT, Biotechnology)与信息技术(IT, Information Technology),在疫苗设计策略、序列建模及功能验证等关键环节持续攻关,显著提升了疫苗的靶向性与稳定性,为顺利开展临床试验奠定了坚实基础。”在抗原设计阶段,研发团队深入研究了FIPV病毒的感染与免疫机制,利用谢震教授团队自主研发的RNA序列人工智能模型,对mRNA疫苗进行了序列优化,并通过动物免疫攻毒实验锁定了病毒核心靶点。此外,谢震教授与两家生物科技公司密切合作,实现了mRNA生产原料和递送系统的国产化替代,大幅降低生产成本,推动该产品从实验室向临床应用的转化,有望将全球首个猫传染性腹膜炎mRNA疫苗产品推向国内外市场,助力中国跻身掌握该核心技术的少数动物保健品国家行列。


谢震教授是医学合成生物学领域的领先专家,他将控制理论、信息技术与生物医学相结合,重点聚焦在人工生物元件、线路设计优化,开发可编程的基因药物,用于肿瘤治疗和传染病防治。在RNA疫苗的开发方面,谢教授带领团队取得了重要突破,尤其在宠物医疗领域的应用上填补了全球空白。这一科研成果的取得,离不开扎实的科学理论支持和前沿的技术应用。谢震教授主编的《医学合成生物学》一书,系统介绍了合成生物学在医学中的发展和应用,是了解该领域未来发展不可或缺的宝贵学术资料。该书从理论基础到前沿技术,全面阐述了医学合成生物学的发展脉络与应用前景,对于科研人员、临床医生及对生物技术感兴趣的读者,都是一份重要参考。强烈推荐大家阅读这本书,谢震教授带您深入了解医学合成生物学的广泛应用和前景。

信息来源:GutSir
⽑润泽
助理教授,特别研究员,博士生导师
清华大学 深圳国际研究生院生物医药与健康工程研究院
教育及⼯作经历
2024-08 ⾄今 助理教授、特聘研究员 | 清华⼤学
医药与健康⼯程研究院,清华⼤学深圳国际研究⽣院(iBHE, SIGS)
2021-01 ⾄ 2024-07 博⼠后 | 加州理⼯学院, 美国
导师: Frances H. Arnold 教授 | SNSF 博⼠后奖学⾦
2016-07 ⾄ 2020-12 博⼠ | 化学 | 瑞⼠联邦理⼯学院(洛桑),瑞⼠
导师: 胡喜乐(Xile Hu)教授 | 优秀博⼠论⽂
2013-09 ⾄ 2016-07 硕⼠ | 化学⽣物学 | 北京⼤学
导师: 叶新⼭ 教授 | 优秀毕业⽣
2009-09 ⾄ 2013-07 学⼠ | 化学 | 河南师范⼤学
导师: 渠桂荣 教授, 郭海明 教授 | 优秀毕业⽣
研究方向
⽣物催化, 蛋⽩⼯程, 定向进化
代表性论文
1. Liang, L.; Wang, Y. H.; Cui, C. X.; Deng, X. S.; Wang, S. L.; Guo, H. M.; Li, Y.; Niu, H. Y.; Mao, R.* NADH AnaloguesEnable Metal- and Light-Free Decarboxylative Functionalization. Angew. Chem. Int. Ed. 2024, e20241513
2. Mao, R.; Gao, S.; Qin, Z.; Wu, S. J.; Li, Z. Q. Arnold, F. H. Biocatalytic, Enantioenriched Primary Amination ofTertiary C–H Bonds. Nat. Catalysis. 2024, 7, 585.
3. Mao, R.; Arnold, F. H. An Engineered Enzyme Enables Stereoconvergent Alkylation of Alkene Mixtures. Nat.Synth. 2024, 3, 160.
4. Mao, R.; Taylor, D. M.; Wackelin, D. J.; Wu, S. J.; Sicinski, K. M.; Arnold, F. H. Biocatalytic, StereoconvergentAlkylation of (Z/E)-Trisubstituted Silyl Enol Ethers. Nat. Synth. 2024, 3, 256.
5. †Wackelin, D. J.; †Mao, R. (共同⼀作); Sicinski, K. M.; Zhao, Y.; Chen, K.; Arnold, F. H. Enzymatic Assembly ofDiverse Lactone Structures: An Intramolecular C–H Functionalization Strategy. J. Am. Chem. Soc. 2024, 146,1580. (†同等贡献).
6. Mao, R.; Wackelin, D. J.; Jamieson, C. S.; Rogge, T.; Gao, S.; Das, A.; Taylor, D. M.; Houk, K. N.; Arnold, F. H.Enantio- and Diastereoenriched Enzymatic Synthesis of 1,2,3-Polysubstituted Cyclopropanes from (Z/E)-Trisubstituted Enol Acetates. J. Am. Chem. Soc. 2023, 145, 16176.
7. Mao, R.; Bera, S.; Turla, A. C.; Hu, X. Copper-Catalyzed Intermolecular Functionalization of Unactivated C(sp3)– H Bonds and Aliphatic Carboxylic Acids. J. Am. Chem. Soc. 2021, 143, 14667.
8. †Bera, S.; †Mao, R. (共同⼀作); Hu, X. Enantioselective C(sp3)–C(sp3) Cross-Coupling of Non-Activated AlkylElectrophiles via Nickel Hydride Catalysis. Nat. Chem. 2021, 13, 270. (†同等贡献).
9. Mao, R.; Bera, S.; Cheseaux, A.; Hu, X. Deoxygenative Trifluoromethylthiolation of Carboxylic Acids. Chem. Sci.2019, 10, 9555.
10. Mao, R.; Balon, J.; Hu, X. Decarboxylative C(sp3)–O Cross-Coupling. Angew. Chem., Int. Ed. 2018, 57, 13624.
电⼦邮箱: 该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
谭英
副教授,博士生导师
清华大学 深圳国际研究生院
教育工作经历
2012年10月至今, 清华大学深圳国际研究生院,讲师、副教授
2016年10月-2017年10月,新加坡国立大学,访问学者
2010年07月-2012年07月,清华大学,博士后
2005年09月-2010年07月,华中师范大学/南开大学,理学博士
2001年09月-2005年07月,华中师范大学,理学学士
研究方向
围绕肿瘤精准诊疗,建立一系列无创、高灵敏、高特异的疾病诊断体系和装置;发展基于细胞表型、合成生物学和生物正交的药物开发体系,调控和干预生命过程;开发全新的人工智能算法,并应用于多组学研究、疾病诊断和药物设计。
所获荣誉
2021年深圳市优秀教师
清华大学第九届青年教师教学大赛三等奖
清华大学优秀硕士学位论文指导教师
代表性论文
该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
李斐然
助理教授,特别研究员,博士生导师
清华大学 深圳国际研究生院生物医药与健康工程研究院
教育工作经历
2023年-至今, 清华大学深圳国际研究生院,助理教授,博士生导师
2021年-2023年,查尔姆斯理工大学,博士后(合作导师:Jens Nielsen院士)
2017年-2021年,查尔姆斯理工大学,系统生物学专业,博士(导师:Jens Nielsen院士)
2014年-2017年,天津大学,生物化工专业,硕士(导师:赵学明教授)
2010年-2014年,天津师范大学,化学生物学专业,学士
研究方向
致力于通过开发新方法和新技术分析生物大数据和研究生物系统,以构建数字生命,揭示内在机理,促进合成生物学和生物医药研究。研究涉及计算生物学、系统生物学、机器学习、化学、药物代谢等多个领域。
主要有以下三个研究方向:
面向医药健康,开发并分析哺乳动物细胞、器官及人体的数字孪生代谢模型(数字孪生人类)。
探索细胞代谢暗物质,促进新途径和新酶的发现,加速生物合成途径设计及潜力挖掘(数字细胞)。
开发深度学习模型,助力解析蛋白序列、功能及其参数间的关系。”
所获荣誉:
代表性论文
# Co-first author, * Corresponding author
其他相关发表论文:
Google scholar:https://scholar.google.com/citations?user=Zn6Gy-IAAAAJ&hl=en
ORCID: https://orcid.org/0000-0001-9155-5260
该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
深圳市南山区西丽深圳大学城清华校区国际一期A栋1603
张灿阳
副教授,研究员,博士生导师
清华大学 深圳国际研究生院学院
教育工作经历
2024.01至今 清华大学深圳国际研究生院,副教授,博士生导师
2021.05-2023.12 清华大学深圳国际研究生院,助理教授,博士生导师
2019.06-2020.12 麻省理工学院新加坡科研中心,博士后
2016.08-2019.05 华盛顿州立大学,博士后
2015.03-2016.07 国家纳米科学衷心,助理研究员
2012.10-2013.10 慕尼黑大学,联合培养
2009.09-2014.12 华南理工大学,硕博连读
研究方向
设计并开发新型靶向递送体系与技术,创制功能超越的工程化生物-材料复合体系,实现生物体系的功能调控,拓展生物-材料复合体在生物医药与健康领域中的应用。已发表第一/通讯作者48篇SCI论文,包括Science Advances,Advanced Materials,AIChE Journal,Chemical Engineering Science等,封面文章和ESI高被引论文4篇,引用3500余次,H-因子33,担任学术期刊《Health Engineering》(清华大学出版社)执行主编、《Journal of Future Foods》编委、《Exploration》学术编辑。
所获荣誉:
广东省“珠江人才计划”(2022)
深圳市“海外高层次人才计划”(2021)
清华大学年度教学优秀奖(2023)
代表性论文
邮箱:该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
电话:13522067013
王润铭
助理教授,特别研究员,博士生导师
清华大学 深圳国际研究生院生物医药与健康工程研究院
工作经历
02/2022 – 至今 助理教授、特别研究员、博士生导师,生物医药与健康工程研究院,深圳国际研究生院,清华大学
10/2018 – 10/2021 博士后研究员,理学院化学系,香港大学
03/2018 – 9/2018 科研助理,理学院化学系,香港大学
教育经历
01/2014 – 06/2018 香港大学理学院化学系,博士 (生物无机化学)
导师: 孙红哲叶志成范港喜基金教授(生物无机化学)、讲座教授,理学院化学系,香港大学
共同导师: 高一村副教授,李嘉诚医学院微生物学系,香港大学
研究方向
金属化学生物学、合成生物学、合成化学和计算生物学等领域的交叉研究。围绕生物资源的多尺度挖掘与生物分子的金属化活性改造,设计创新跨物种金属蛋白肽元件库,开发数据驱动的金属蛋白肽设计与定向进化平台技术,定制功能化金属基抗感染、抗肿瘤及纤维化药物及慢病防控产品。
所获荣誉
日内瓦国际发明展金奖(2023)、英国皇家化学会道尔顿探索奖(2023)、《麻省理工科技评论》“35岁以下科技创新35人” 亚太区 (2022)、国家海外高层次人才(青年)项目(2022)、 “鹏城孔雀计划”特聘岗位B类人才 (2022)、香港十大创科新闻 (2021)、香港大学知识交流奖 (2021)、香港大学-巴斯德所研究极奖学金(唯二) (2021)、AsBIC9最佳展示奖 (2018)、英国皇家化学会书籍奖 (2016)
代表性论
1. Wang R#, Lai TP#, Gao P#, Zhang H#, Ho PL, Woo PC, Ma G, Kao RYT, Li H, Sun H*. “Bismuth antimicrobial drugs serve as broad-spectrum metallo-β-lactamase inhibitors” Nat. Commum., 2018, 9, 439.
2. Sun H#*, Zhang Q#, Wang R#*(Co-first author and Co-corresponding author), Wang H, Wong YT, Wang M, Hao Q, Yan A, Kao YT, Ho PL, Li H, “Resensitizing superbugs carrying both blaMBL and mcr genes to antibiotics by auranofin” Nat. Commum., 2020, 11, 5263.
3. Yuan S#, Wang R#(Co-first author), Chan FW#, Zhang J, Cheng T, Chik KH, Ye Z-W, Wang S, Jin L, Li H, Jin DY, Yuen KY*, Sun H*, “Metallodrug ranitidine bismuth citrate suppresses SARS-CoV-2 replication and relieves virus-associated pneumonia in Syrian hamsters” Nat. Microbiol, 2020, 5, 1439–1448.
4. Cheng H, Huang S, Cai L., Xu Y, Wang R*, Zhang Y.*., “Focus Your Attention: Multiple Instance Learning with Attention Modification for Whole Slide Pathological Image Classification.” IEEE Transactions on Circuits and Systems for Video Technology. 2025, DOI: 10.1109/TCSVT.2025.3528625.
5. Zhang Q#, Wang R#(Co-first author), Wang M, Liu C, Koohi-Moghadam M, Wang H, Ho P-L, Li H, Sun H*, Proc. Natl. Acad. Sci. U.S.A., 2022, 119, e2119417119.
邮箱:该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
马少华
清华大学 清华大学深圳国际研究生院
长聘副教授,博士生导师
教育工作经历
2021-01 至 今, 清华大学深圳国际研究生院, 生物医药与健康工程研究院, 副教授
2020-08 至 2020-12, 清华-伯克利深圳学院, 精准医学与公共健康研究中心, 副教授
2017-08 至 2020-07, 清华-伯克利深圳学院, 精准医学与公共健康研究中心, 研究员
2013-11至 2017-08, 英国牛津大学,博士后,导师:Hagan Bayley
2010-10 至 2013-10, 剑桥大学, 化学, 博士;Wilhelm Huck和Chris Abell
2009-10 至 2010-09, 剑桥大学, 化学, 硕士;Wilhelm Huck
2005-09 至 2009-07, 中山大学, 高分子材料与工程, 学士,导师:陈旭东
研究方向
类器官合成生物制造技术与应用研究
所获荣誉:
(1) 马少华(1/7); 日内瓦国际发明特别展金奖, 世界知识产权组织(WIPO), 发明, 国际学术奖, 2022(马少华; 赵浩然; 蔡咏德; 李佳楠; 李家炜; 曹远雄; 杨浩威)
代表性论文
[1]. Ma, Shaohua*; Wang, Wanlong; Zhou, Jiaqi; Liao, Shangfeng; Hai, Cheng; Hou, Yibo; Zhou, Zhichun; Wang, Zitian; Su, Yingshi; Zhu, Yu; Dai, Xiaoyong; Zhao, Yuan; Liao, Sha; Cai, Yongde*; Xu, Xun*; Lamination-based organoid spatially resolved transcriptomics technique for primary lung and liver organoid characterization, PNAS, 2024, 121, e2408939121.
[2]. Yang, Haowei; Li, Jiawei; Zheng, Yichao; Cao, Yuanxiong; Zhu, Yu; Sang, Gan; Galan, Edgar A; Ruan, Chuqian; Ma, Wilson M. J.; Dai, Xiaoyong; Cai, Yongde*; Ma, Shaohua*; Ultra-small tissue-compatible organoid printer for rapid and controllable modeling of respiratory organoids, Device (Cell Press), 2024, 2, 100420.
[3]. Zheng, Yichao; Han, Fei; Wu, Zhengyu; Wang, Bingjie; Chen, Xingchi; Boulouis, Caroline; Jiang, Yuebin; Ho, Amanda; He, Dan; Sia, Wan Rong; Mak, Jeffrey Y.W.; Fairlie, David P.; Wang, Lin-Fa; Sandberg, Johan K.; Lobie, Peter E.; Ma, Shaohua*; Leeansyah, Edwin*; MAIT cell activation and recruitment in inflammation and tissue damage in acute appendicitis, Science Advances, 2024, 10, eadn6331.
[4]. Zheng, Yichao; Tian, Qinyong; Yang, Haowei; Cai, Yongde; Zhang, Jiaxin; Wu, Yifen; Zhu, Shuo; Qiu, Zuocheng; Lin, Yimin; Hong, Jiangquan; Zhang, Yi; Dockrell, David; Ma, Shaohua*; Identification of nicotinic acetylcholine receptor for N-acetylcysteine to rescue nicotine-induced injury using beating cilia in primary tissue derived airway organoids, Advanced Science, 2024, 11, 2407054.
[5]. Zhou, Jiaqi; Huang, Yi-chun; Wang, Wanlong; Li, Jiawei; Hou, Yibo; Yi, Ziqi; Yang, Haowei; Hu, Keer; Zhu, Yu; Wang, Zitian; Ma, Shaohua*; Chronotoxici-plate containing droplet-engineered rhythmic liver organoids for drug toxicity evaluation, Advanced Science, 2024, 11, 2305925.
[6]. Liu, Hanghang; Xu, Haohan; Zhu, Yu; Wang, Zitian; Hu, Danni; Yang, Lingxiao; Zhu, Yinheng; Galan, Edgar A; Huang, Ruqi; Peng, Haiying; Ma, Shaohua *; A large model-derived algorithm for complex organoids with internal morphogenesis and digital marker derivation, Analytical Chemistry, 2024, 96, 19258-19266.
[7]. Feng, Yilin; Fan, Jiaqi; Cheng, Yifan; Dai, Qionghai; Ma, Shaohua*; Stress regulates Alzheimer's disease progression via selective enrichment of CD8+ T cells, Cell Reports, 2023, 42, 113313.
[8]. Cao, Yuanxiong; Tan, Jiayi; Zhao, Haoran; Deng, Ting; Hu, Yunxia; Zeng, Junhong; Li, Jiawei; Cheng, Yifan; Tang, Jiyuan; Hu, Zhiwei; Hu, Keer; Xu, Bing; Wang, Zitian; Wu, Yaojiong; Lobie, Peter E; Ma, Shaohua*; Bead-jet printing enabled sparse mesenchymal stem cell patterning augments skeletal muscle and hair follicle regeneration, Nature Communications, 2022, 13, 7463.
[9]. Jiang, Shengwei; Zhao, Haoran; Zhang, Weijie; Wang, Jiaqi; Liu, Yuhong; Cao, Yuanxiong; Zheng, Honghui; Hu, Zhiwei; Wang, Shubin; Zhu, Yu; Wang, Wei; Cui, Shuzhong; Lobie, Peter E.; Huang, Laiqiang*; Ma, Shaohua*; An automated organoid platform with inter-organoid homogeneity and inter-patient heterogeneity, Cell Reports Medicine, 2020, 1, 100161.
[10]. Yong Wang, Mengqi Ji, Shengwei Jiang, Xukang Wang, Jiamin Wu, Feng Duan, Jingtao Fan, Laiqiang Huang, Shaohua Ma*, Lu Fang*, Qionghai Dai*, Augmenting vascular disease diagnosis by vasculature-aware unsupervised learning, Nature Machine Intelligence, 2020, 2, 337-346.
电话:13827431329
电子邮箱:该Email地址已收到反垃圾邮件插件保护。要显示它您需要在浏览器中启用JavaScript。
地址:深圳市南山区西丽大学城清华校区国际一期A1706